Arbeitsgruppe Cambridge: Genetische Resilienz bei neurodegenerativen Krankheiten
Für neurodegenerative Pathologien, wie z.B. Schlaganfälle, Alzheimers‘ oder Rückenmarksverletzungen, gibt es trotz intensivster Forschungsbemühungen weiterhin keine zufriedenstellenden Behandlungsstrategien. Dies liegt unter anderem daran, dass umfassende genetische Netzwerkanalysen, wie sie zu Hunderten bei Krebszellen gemacht wurden und somit Grundlagen für die erfolgreichen neuen Therapien darstellen, bei Nervenzellen technisch fast unmöglich sind. Nach jahrelanger Forschungsarbeit ist es uns gelungen, ein extrem effektives Verfahren für siRNA-vermittelten Knockdown in vitro zu entwickeln, mit dem jedes einzelne Gen in Nervenzellen akut ausgeschalten und der daraus resultierende zelluläre Phänotyp untersucht bzw. quantifiziert werden kann.
Mit diesem Verfahren können wir die zugrundeliegenden genetischen Netzwerke eines pathologischen Phänotyps detailliert beschreiben. Dabei erwarten wir, dass es einige Gene gibt, deren siRNA-Knockdown den Phänotyp noch weiter verstärken wird, aber auch manche Gene, deren Knockdown den pathologischen Phänotyp reduziert, d.h. kompensieren kann. Unser Ziel ist es, ‚kompensatorische Gene‘ für verschiedenste neurodegenerative Krankheiten zu identifizieren und diese dann als Ziele für die Medikamentenentwicklung bereitzustellen.
Forschung anderer Labore hat gezeigt, dass selbst letale Mutationen bei einigen wenigen Menschen keinen Phänotyp hervorrufen, d.h. deren individuelles genetisches Netzwerk konnte die Mutation kompensieren (Chen at al., Nature Biotechnology, 2016). Mit diesem Ansatz der genetischen Resilienz wollen wir die zwei größten Herausforderungen neuronaler Degeneration angehen: 1. Reduktion von neuronalen Proteinaggregationen bei Krankheiten wie Alzheimer, Parkinson, ALS, etc. und 2. Verbesserung neuronaler Regeneration nach Schlaganfällen, Rückenmarksverletzungen, etc.
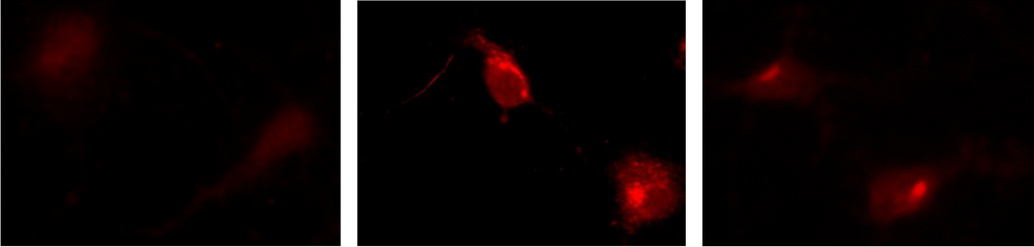
Kandidat für kompensatorisches Gen dessen siRNA-Knockdown neuronale Proteinaggregation um ca. 50% reduziert.
Links: Kontroll-Nervenzellen zeigen nur Hintergrunds-Fluoreszenz
Mitte: Transgene Nervenzellen mit Proteinaggregaten
Rechts: Proteinaggregation in transgenen Nervenzellen war nach siRNA-Knockdown eines potentiellen kompensatorischen Gens deutlich reduziert
Erste kleine Machbarkeitsstudien mit siRNAs gegen 45 ausgesuchte Gene waren sehr überzeugend und haben gezeigt, dass mit unserem Verfahren kompensatorische Gene identifiziert werden können. Im nächsten Schritt soll das Verfahren auf die etwa 800 Gene des „druggable genoms“ (Gene mit FDA-genehmigten Wirkstoffen/Medikamenten) ausgeweitet werden. Der Effekt möglicher kompensatorischer Gene auf den Phänotyp kann dann umgehend mit den bekannten Wirkstoffen getestet werden. Mittelfristig sollen Genom-weite Hochdurchsatz-Verfahren durchgeführt werden, um möglichst alle kompensatorischen Gene zu identifizieren und das genetische Netzwerk umfassend zu analysieren. Natürlich soll das Verfahren auch dafür eingesetzt werden, um wesentliche neuronale Prozesse und Strukturen detailliert genetisch zu untersuchen, wie z.B. Neurogenese oder Synaptogenese. [Das von uns entwickelte Hochdurchsatz-Verfahren ist aus patentrechtlichen Gründen noch nicht publiziert.]
Wir freuen uns über Bewerbungen für Masterarbeiten, sowie für (medizinische) Doktorarbeiten.